Auswertung von räumlichen Daten
- Einleitung
- Daten herunterladen
- Datei mit dem raster package einlesen
- Daten plotten
- Teilbereiche Ausschneiden
- Resampling
- Abhängigkeit zwischen Höhe und NDVI im Streudiagramm
Einleitung
Dieses Tutorial soll zeigen, wie man in R mit räumlichen Daten arbeitet. Dabei werden wir das Package raster verwenden. Als Daten werden wir sogenannte NDVI-Daten verwenden. Die Abkürzung NDVI steht für Normalized Difference Vegetation Index. Es handelt sich hier um Daten die von einem Satelliten, betrieben durch das NASA/Goddard Space Flight Center aufgenommen werden. Der NDVI wird aus dem Infrarotanteil (\(IR\)) und dem Rotanteil (\(R\)) der Satellitenbilder berechnet:
Das Ergebnis sind Bilder, die besonders gut den Bedeckungsgrad an Vegetation und deren Vitalität wiedergeben. Der NDVI wird verwendet, um Trockenheit zu überwachen, landwirtschaftliche Produktion zu überwachen und zu planen, Zonen mit Brandgefahr zu erkennen und das Vordringen der Wüste kartografisch darzustellen.1
In diesem Tutorial verwenden wir den durch Moderate Resolution Imaging Spectroradiometer (MODIS) normalisierte Difference Vegetation Index (NDVI), der durch die Global Inventory Modeling and Mapping Studies (GIMMS) Group des NASA/Goddard Space Flight Center aufgenommen und verarbeitet wurde, sowie finanziell unterstützt wurde vom Global Agricultural Monitoring project der USDA’s Foreign Agricultural Service (FAS). Die Daten können unter https://gimms.gsfc.nasa.gov/MODIS/std/GMOD09Q1/tif/NDVI/ abgerufen werden.
Die Daten liegen im GeoTIFF-Format vor. Messungen von 8 Tagen werden jeweils zu einer Datei zusammen gefasst. Der tatsächliche Messzeitpunkt für jeden Pixel wird seit 2012 in einem zweiten Raster gespeichert. Dies soll aber hier vernachlässigt werden und stattdessen nur der Beginn der 8-Tages-Periode verwendet werden.
Die folgende Readme-Datei gibt Auskunft über die Formatierung der Daten und die Konvention für die Dateinamenvergabe: https://gimms.gsfc.nasa.gov/MODIS/README.txt
Daten herunterladen
Die Daten befinden sich in Unterordnern zu der oben genannten URL. Der erste Unterordner definiert das Jahr (z.B. 2001), der zweite Unterordner den Tag des Jahres (z.B. 049 für den 49. Tag). In diesem Ordner liegen dann die Dateien, die ähnlich wie diese Datei benannt sind: GMOD09Q1.A2010001.08d.latlon.x00y02.6v1.NDVI.tif
Dabei bedeuten laut der Readme-Datei die einzelnen Teile des Dateinamen folgendes:
Qualifier Value Description
--------- ----- -----------
Dataset GMOD09Q1 GIMMS MODIS Terra (MOD=Terra MYD=Aqua)
Start date A2010001 Starting year 2010, day-of-year 001
Composite period 08d 8-day composite
Projection latlon Lat/Lon grid
9x9 Tile index x00y02 Column 00, Row 02
Versions 6v1 MODAPS collection 6, GIMMS version 1
Layer name NDVI Normalized Vegetation Index
File format tif Tagged Image File
Den 9x9 tile index kann man nach der Readme-Datei wie folgt berechnen:
Since the size of a global grid file may be too large for users,
the grid is divided into 40 columns and 20 rows (starting at the
upper left corner at 180W 90N) to create 9x9 degree tiles of
4000 x 4000 pixels.
Tiles are identified by {x,y} indexes and labeled in the filename
(5th qualifier).
To calculate the {x,y} tile index for a given {Lon,Lat}:
x = floor((180 + Lon) / 9)
y = floor(( 90 - Lat) / 9)
Wir wählen uns eine Datei aus, auf der Mitteleuropa zu sehen ist und verwenden den Datensatz von 2001 vom 209. Tag des Jahres, also einem Tag Ende Juli. Die url zu dieser Datei ist folgende:
url <- "https://gimms.gsfc.nasa.gov/MODIS/std/GMOD09Q1/tif/NDVI/2001/209/GMOD09Q1.A2001209.08d.latlon.x21y04.6v1.NDVI.tif.gz"Diese laden wir nun herunter und entpacken sie:
# Komprimierte Datei runterladen
download.file(url, basename(url), method="curl", quiet=T)
# Datei enptacken
filename <- R.utils::gunzip(basename(url), overwrite=T)Zum entpacken der .gz Dateien muss das Paket R.utils installiert sein. Falls dies noch nicht installiert ist einfach vorher install.packages("R.utils") ausführen.
Datei mit dem raster package einlesen
Die heruntergeladenen Datei wollen wir nun mit dem raster package in R verarbeiten.
Laden wir also das Paket:
library(raster)Mit diesem Befehl laden wir die .tif-Datei als raster in R:
NDVI_raster <- raster(filename)Daten plotten
plot(NDVI_raster)Etwas ist hier noch falsch! Aus der Readme-Datei können wir entnehmen, dass alle Werte über 250 keine tatsächlichen NDVI-Daten darstellen und dass wir die Werte darunter noch umrechnen müssen:
The valid range of NDVI, 8-bit layers is [0 - 250].
Values ranging [251 - 255] are reserved for mask values:
Value Description
----- -----------
251 (empty)
252 (empty)
253 Invalid land (out of range NDVI)
254 Water
255 No data (unfilled, cloudy, or snow contaminated)
To convert from NDVI 8-bit unsigned integer [0 - 250] to
floating-point [0.0 - 1.0]:
<FLOAT NDVI> = <UINT8 NDVI> x 0.004
Wir definieren deshalb eine Funktion, die alle Werte über 250 durch NA ersezt und alle darunter mit 0.004 multipliziert:
remap <- function(x){
x[x>250] <- NA
x <- x * 0.004
return(x)
}Mit dieser Funktion können wir nun das Raster umrechnen. Dazu benutzen wir die Funktion calc() aus dem raster package und übergeben als Argument fun unsere Funktion remap. Damit wir die Umrechnung beim nächsten mal nicht erneut ausführen müssen, speichern wir das umgerechnete Raster in einer Datei mit dem Namen "NDVI_raster_remapped.tif".
NDVI_raster_remapped <- calc(NDVI_raster, fun=remap, filename="NDVI_raster_remapped.tif", overwrite=TRUE)Diese Datei können wir dann später direkt mit dem raster() Befehl laden, wie oben mit der Originaldatei gezeigt.
Plotten wir nun unser umgerechnetes Raster:
plot(NDVI_raster_remapped)Die NDVI-Werte reichen nun von 0 bis 1. Zusätzlich sieht man nun, dass gebietsweise Daten fehlen und weiß angezeigt werden.
Teilbereiche Ausschneiden
Wir wollen nur einen kleinen Ausschnitt aus dieser Karte verwenden. Diesen Ausschnitt können wir zum Beispiel mit der Funktion drawExtent() direkt mit der Maus aus einem bestehenden Plot auswählen:
ext <- drawExtent(show = T)Oder wir definieren den Ausschnitt mit vier Koordinaten:
ext <- extent(10,11, 46,47)Egal auf welche Weise wir ext definiert haben können wir damit eine Karte plotten, die nur diesen Ausschnitt enthält:
plot(NDVI_raster_remapped, ext = ext)Hier verwenden wir ext zuerst nur, um einen Kartenausschnitt darzustellen, nicht um unsere Daten tatsächlich zu beschneiden.
Wir können auch den extent einer anderen Karte verwenden, um unsere Karte zuzuschneiden. Laden wir dazu z.B. ein digitales Geländemodell (DGM, engl. DEM) von viewfinderpanoramas.org herunter:
elevURL <- "http://viewfinderpanoramas.org/dem1/n46e010.zip"
# Zip-Datei runterladen
download.file(elevURL, basename(elevURL))
# Datei entpacken
elevFile <- unzip(basename(elevURL))Die heruntergeladenen Daten laden wir wieder mit dem raster-package:
elevation <- raster(elevFile)Dieses Raster benutzen wir nun, um unser NDVI-Raster zuzuschneiden:
NDVI_raster_remapped_crop <- crop(NDVI_raster_remapped, elevation)Resampling
Wenn wir uns das digitale Höhenmodell anschauen, fällt sofort eine Ähnlichkeit mit dem räumlichen Muster im NDVI-Bild auf.
plot(elevation, col=terrain.colors(100))Daher kann man hier eine Abhängikgeit des NDVI von der Höhenlage vermuten. Der gewählte Ausschnitt liegt in den Alpen mit Höhenlagen bis über 3000m, also Höhen, in denen irgendwann gar keine Vegetation mehr zu finden ist. Dazwischen gibt es wohl einen fließenden Übergang. Diesen wollen wir nun zeigen.
Um die Abhängigkeit des NDVI von der Geländehöhe zu zeigen müssen wir jedem Wert aus dem NDVI-Raster einen Wert aus dem DGM-Raster zuweisen. Da wir das eine Raster mit dem anderen zugeschnitten haben, haben sie grob (nicht exakt) die gleiche Ausdehnung. Allerdings haben sie nicht die gleiche Auflösung:
elevation## class : RasterLayer
## dimensions : 3601, 3601, 12967201 (nrow, ncol, ncell)
## resolution : 0.0002777778, 0.0002777778 (x, y)
## extent : 9.999861, 11.00014, 45.99986, 47.00014 (xmin, xmax, ymin, ymax)
## crs : +proj=longlat +datum=WGS84 +no_defs
## source : N46E010.hgt
## names : N46E010
## values : -32768, 32767 (min, max)NDVI_raster_remapped_crop## class : RasterLayer
## dimensions : 445, 445, 198025 (nrow, ncol, ncell)
## resolution : 0.00225, 0.00225 (x, y)
## extent : 9.999, 11.00025, 45.999, 47.00025 (xmin, xmax, ymin, ymax)
## crs : +proj=longlat +datum=WGS84 +no_defs
## source : memory
## names : NDVI_raster_remapped
## values : 0, 1 (min, max)Dies lässt sich beheben, indem wir die Funktion resample() verwenden. In diesem Falle berechnen wir die Auflösung des höher aufgelösten Rasters auf die des geringer aufgelösten um. Das DGM ist in diesem Falle höher aufgelöst und das NDVI-Raster ist das mit der geringeren Auflösung. Es würde auch anders herum funktionieren, allerdings müsste R dafür interpolieren. Wir wollen aber nicht künstlich Informationen erzeugen, wo eigentlich keine sind. Daher aggregieren wir lieber die Höheninformationen zu einem geringer aufgelösten Raster:
elevation_resampled <- resample(elevation, NDVI_raster_remapped_crop)Schauen wir uns das neu gesampelte Raster an, wird deutlich, dass es nun exakt die gleiche Auflösung und exakt den gleichen extent wie das NDVI-Raster hat:
elevation_resampled## class : RasterLayer
## dimensions : 445, 445, 198025 (nrow, ncol, ncell)
## resolution : 0.00225, 0.00225 (x, y)
## extent : 9.999, 11.00025, 45.999, 47.00025 (xmin, xmax, ymin, ymax)
## crs : +proj=longlat +datum=WGS84 +no_defs
## source : memory
## names : N46E010
## values : 231.8971, 3839.372 (min, max)Nun können wir jeder Zelle aus dem einen Raster exakt eine Zelle aus dem anderen Raster gegenüberstellen.
Abhängigkeit zwischen Höhe und NDVI im Streudiagramm
Um jeder Zelle aus dem einen Raster eine Zelle aus dem anderen Raster gegenüber zu stellen verwenden wir den Befehl getValues(). Dieser formt das Raster in einen Vektor aus Werten um. Wir erzeugen nun einen data.frame, der die Zellwerte aus den beiden Rastern zeilenweise gegenüber stellt:
scatterData <- data.frame(
elev = getValues(elevation_resampled),
NDVI = getValues(NDVI_raster_remapped_crop)
)Diesen data.frame können wir nun nutzen um einen scatterplot darzustellen:
plot(scatterData, pch=20, cex=0.2, xlab="elevation [m]", ylab="NDVI", col=rgb(0,0,0,0.1))
abline(v=2000, lty=4)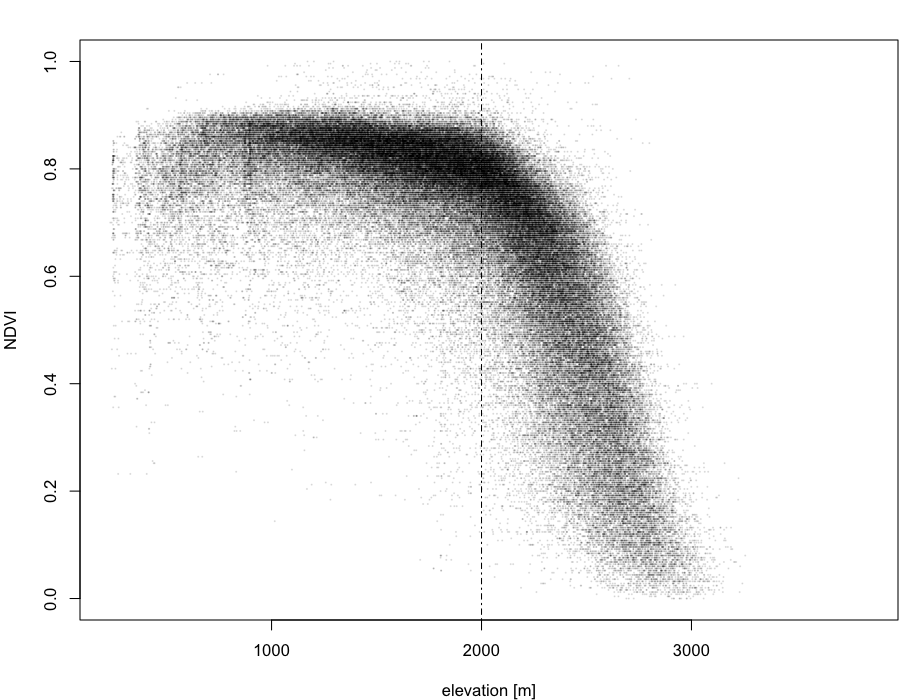
Man erkennt sofort eine Gesetzmäßigkeit in der Punktwolke: Bis etwa 2000m bleibt der NDVI weitestgehend konstant, hier besteht keine Abhängigkeit zur Höhe. Ab 2000m besteht eine ungefähr lineare Abhängigkeit zur Höhe: der NDVI sinkt mit steigender Höhe, bis er auf etwa 2900m im mittel Null ist. Um die 2000m herum könnte man die Abhängigkeit auch mit einer Kurve beschreiben. Auf den letzten Höhenmetern bis zur Obergrenze scheint die Abhängigkeit noch einmal in die andere Richtung abzuknicken. In allen Bereichen streuen die Werte natürlich um einen Mittelwert herum.
An dieser Stelle könnte man natürlich mit weiteren analysen ansetzen. Man könnte versuchen die ersichtliche Abhängigkeit stückweise linear zu beschreiben (z.B. mit lm(). Oder man versucht eine Kurve anzupassen (z.B. mit smooth.spline()). Desweiteren könnte man die NDVI auch auf eine Abhängigkeit zur Hangausrichtung (aspect) und zujm Gefälle (slope) hin untersuchen. Hier ist der Befehl terrain() aus dem raster-package nützlich, mit dem man slope und aspect berechnen kann. Dies sollen jedoch Themen für weitere Tutorials bleiben. Oder Sie probieren sich einfach selber daran.
Wie geht’s weiter? Als Fortsetzung zu dieser Übung empfiehlt sich die Übung zu RasterStacks.
-
http://desktop.arcgis.com/de/desktop/latest/manage-data/raster-and-images/ndvi-function.htm ↩