Empirische Verteilungsfunktionen ableiten
Dies ist die dritte Übung aus einer Reihe von Übungen zu hydrologisch/metereologischen Datensätzen und deren Auswertung. Sie setzt die Übung zur Grasreferenzverdunstung fort.
In dieser Übung werden wir mit der Funktion calcET0 aus der letzen Übung weiter arbeiten. Um eine funktionierende Version der Funktion zu erhalten kann die “master”-Version des RgetDWDdata package installiert werden:
library(devtools)
install_github("nFrechen/RgetDWDdata", ref="master")In dieser Version enthält das Paket zusätzlich Hilfeseiten zu den Themen:
help(calcET0)help(evap.net.radiation)undhelp(es)
Theorie
Von der Deterministik in die Statistik
In der Deterministik kann einer bestimmten Kombination aus Eingangsparametern jeweils ein bestimmtes Ergebnis, ein bestimmter Output zugeordnet werden. Ein deterministisches Modell kommt bei gleichen Eingabeparametern immer zu dem gleichen Ergebnis. Manche Prozesse in der Natur, wie z.B. die Planetenbewegungen können wir durch ein solches deterministisches Modell beschreiben, die entsprechenden Parameter exakt messen und deshalb dann z.B. auch die Planetenbewegungen exakt voraus berechnen.
In den Umweltwissenschaften und speziell auch in der Hydrologie haben wir es mit einer Menge Prozesse zu tun,
- die wir A) noch nicht verstehen, was heißt, dass wir sie noch durch kein adäquates Modell beschreiben können und
- B), deren Eingabeparameter, Randbedingungen und Anfangsbedingungen wir nicht genau bestimmen können und vielleicht auch nie genau bestimmen werden können.
Abgesehen von unserem Unwissen über manche Prozesse gibt es mit der Heisenberg’schen Unschärferelation den Beweis, dass wir manches nie simulieren werden können, da es physikalisch unmöglich ist, die Anfangsbedingugnen zu bestimmen, ohne sie zu verändern. Dieses Phänomen begegnet einem vielfach in der Hydrologie: Wir können z.B. mit den gängigen Techniken nicht die Porenraumverteilung einer Bodensäule bestimmen, ohne diese zu zerstören. Würden wir ein Bodenprofil graben um die Verteilung von Makroporen (Risse, Wurzelgänge, Wurmgänge etc.) zu bestimmen, könnten wir damit unser Modell füttern, dieses aber nicht mehr validieren, da wir unser Untersuchungsgebiet zerstört haben.
Warum streuen statistische Werte?
Natürlich folgen statistiche Werte auch Gesetzen, die nach dem Ursache-Wirkungs-Prinzip arbeiten. Aber wenn wir mit statistischen Werten arbeiten können wir keine genauen Aussagen über die Prozesse machen, die zu der entsprechenden Ausprägung eines einzelnen Wertes einer statistischen Variable führen. Wenn es uns z.B. möglich wäre ein einzelnes Wassermolekül zu markieren und zu beobachten wie dies durch eine Bodensäule wandert, wäre es uns trotzdem unmöglich zu berechnen, wann dieses wieder unten aus der Bodensäule heraus kommt, da wir die räumliche Porenverteilung nicht im Detail kennen. Ausserdem wissen wir nicht, wie das Wasserpartikel mit anderen Partikeln interagiert und entsprechend in große oder kleine Poren hineingedrückt wird (und entsprechend schnell oder langsam voran kommt). Mit modernsten Methoden (Röntgen-CT) können wir die räumliche Porenverteilung einer vielleicht 1x1cm Bodensäule bestimmen. Meist wissen wir jedoch nur die statistische Porenraumverteilung aus Siebanalysen. Also können wir auch nur eine statistische Aussage darüber machen, wie schnell die Wasserpartikel in den verschiedenen Poren voran kommen und wie schnell sie am unteren Ende der Säule wieder heraus kommen.
Die Situation ist also folgende:
- Was wir messen ist das Ergebnis einer langen Aneinanderkettung von Einzelprozessen (z.B. Durchfliessen von mehr oder weniger großen Poren).
- Über den Ablauf der einzelnen Prozesse können wir keine Aussagen machen. Entweder ist dies messtechnisch unmöglich oder sogar physikalisch unmöglich (siehe Heisenberg’sche Unschärferelation).
Warum sind statistische Werte nach einer Verteilungsfunktion verteilt?
Trotzdem stellen wir fest, dass die meisten Messvariablen, die einem solchen Prinzip der deterministischen Unbestimmtheit folgen trotzdem statistisch gesehen bestimmten Gesetzmäßigkeiten folgen. Vor allem kann die Streuung der Werte meist durch eine bestimmte Verteilungsfunktion beschrieben werden. Schauen wir uns z.B. die sortierten Werte der an der Station Cottbus gemessenen Temperaturen im Monat Juni an:
Juni <- month(Cottbus$MESS_DATUM)==06
temp <- na.omit(Cottbus$LUFTTEMPERATUR[Juni])
n <- length(temp)
plot(sort(temp), 1:n/n, pch=20)Diese nehmen die typische Form einer S-Kurve an. Diese Kurve lässt sich beschreiben durch die Verteilungsfunktion der Normalverteilung. Die rote Kurve in der nächsten Abbildung beschreibt den Verlauf einer Normalverteilung mit mean=mean(temp) und sd=sd(temp), also Mittelwert und Standardabweichung unserer Stichprobe.
plot(sort(temp), 1:n/n, pch=20)
curve(pnorm(x, mean=mean(temp), sd=sd(temp)), col="red", add=T, lwd=3)Bakannter ist wahrscheinlich die Dichtefunktion der Normalverteilung, die sogenannte Gauß’sche Glockenkurve:
par(mfrow=c(2,1), mar=c(3,4,0.1,0.1))
plot(sort(temp), 1:n/n, pch=20, xlim=c(5,30))
curve(pnorm(x, mean=mean(temp), sd=sd(temp)), col="red", add=T, lwd=3)
legend("topleft", "Verteilungsfunktion", bty="n", cex=1)
plot(density(temp), main="", xlim=c(5,30))
hist(temp, add=T, freq=F)
curve(dnorm(x, mean=mean(temp), sd=sd(temp)), col="red", add=T, lwd=3)
legend("topleft", "Dichtefunktion", bty="n", cex=1)Dargestellt ist hier einmal das Histogramm des Datensatzes (Balken), die mit der Funktion density() ermittelte empirische Dichtefunktion (schwarze Kurve) und die Dichtefunktion der Normalverteilung (mit an den Datensatz angepasstem Mittelwert und Standardabweichung) in rot.
Die Mathematische Beschreibung der Dichtefunktion ist folgende:
\[f(x) = \frac {1}{\sigma\sqrt{2\pi}} e^{-\frac {1}{2} \left(\frac{x-\mu}{\sigma}\right)^2}\]In R ist dies in der Funktion dnorm() implementiert.
Die Verteilungsfunktion ist durch folgende Formel beschrieben:
\[F(x) = \frac{1}{\sigma\sqrt{2\pi}} \int_{-\infty}^x e^{-\frac{1}{2} \left(\frac{t-\mu}{\sigma}\right)^2} \mathrm dt\]Diese ist in R in der Funktion pnorm() implementiert. Andere Verteilungsfunktionen wie die Gamma- bzw. Weibull-Verteilung sind auf ähnliche Weise implementiert. Die Hilfeseite help(Distributions) gibt hier eine Übersicht.
Wie man erkennt ist die Verteilungsfunktion das Integral der Dichtefunktion - sie hat die höchste Steigung, an der Stelle, an der die Glockenkurve ihr Maximum hat. Das Integral der gesamten Glockenkurve ergibt 1, daher geht auch die Verteilungsfunktion von 0 bis 1. Die Schenkel der Normalverteilung setzen sich nach beiden Seiten in die Unendlichkeit fort, daher können wir auch in der Darstellung nicht den Punkt einzeichnen, an dem die Verteilungsfunktion exakt 0 oder exakt 1 erreicht.
Entstehung der Gauß’schen Glockenkurve
Stellen wir uns eine Bodensäule vor, die aus einer zufälligen Verteilung von Poren besteht. In diesem System soll es nur zwei verschiedene Porengrößen geben. Durch die kleinen Poren fließt das Wasser innerhalb von 0.0001 Sekunden, also relativ langsam, durch die großen mit 0.1 Sekunden.
Wir erzeugen nun einen Datensatz, wo 10000 Wasserpartikel durch diese hypothetische Bodensäule fließen und alle verschiedene Wege nehmen. Auf ihrem Weg begegnen sie zufällig verteilt, aber mit gleicher Wahrscheinlichkeit entweder einer großen oder einer kleinen Pore und bewegen sich entsprechend schnell oder langsam fort.
set.seed(1)
n=10000
decisions=1000
X <- rep(0, n)
for(i in 1:n){
for(j in 1:decisions){
X[i] <- X[i] + sample(c(0.0001,0.1), size=1)
}
}Die nächste Grafik zeigt die Verteilung der Zeit, die jedes einzelne Partikel beim Durchfluss durch die Bodensäule gebraucht hat.
par(mar=c(4,4,2,4))
hist(X, freq=F, main=paste(decisions," Entscheidungen bilden 1 Ergebnis, n =", n, "Wiederholungen"), xlab="Ergebnis", breaks=30, font.main=1)
curve(dnorm(x, mean(X), sd(X)), add=T, col="red", lwd=2)Der Vergleich des Histogramms mit der roten Linie zeigt, dass die Messwerte, die auf diese Weise erzeugt wurden sich einer Normalverteilungs-Glockenkurve annähern. Im Mittel brauchen die meisten Partikel etwa 50 Sekunden. Manche Partikel kommen aber schon nach weniger als 45 Sekunden unten an und manche brauchen mehr als 55 Sekunden.
Tatsächlich ist es sogar egal, welche Porenvolumenverteilung wir annehmen, immer kommt eine Normalverteilung heraus. Nur Mittelwert und Standardabweichung der Verteilung ändern sich.
Vorraussetzung für die Bildung dieser Kurve ist, dass die Zufallsentscheidungen unabhängig voneinander sind. Dass es also nicht davon abhängt, wie groß die zuvor durchflossene Pore war, ob als nächstes eine große oder eine kleine Pore durchflossen wird.
Bei Tracerversuchen in realen Bodensäulen ist die Durchbruchkurve keine Gaußkurve, sondern eine sogenannte Schiefe Verteilung: Ein Schenkel (engl: tail) der Verteilung zieht sich weiter nach außen, als der andere.
Solche Verteilungen können z.B. mit der log-normal-Verteilung, der Gamma-Verteilung und der Weibull-Verteilung (siehe Grafik) beschrieben werden. Viele hydrologische Daten weisen eine Schiefe Verteilung auf und können daher mit diesen Verteilungsfunktionen besser beschrieben werden.
Auf der Hilfeseite help(Distributions) findet man einen Überblick über die in R implementierten Verteilungsfunktionen. Andere können über Pakete hinzugeladen werden.
Unterschied zwischen theoretischer und empirischer Verteilungsfunktion
Die Grundgesamtheit ist ein unendlicher Datensatz (\(n\rightarrow\infty\)), den es natürlich nur theoretisch gibt. Die Grundgesamtheit können wir durch eine kontinuierliche Verteilungs- bzw. Dichtefuntion darstellen, die sogenannte theoretische Verteilungfunktion. Wenn wir eine Messkampagne von begrenzter Dauer starten, erzeugen wir natürlich nur eine begrenzte Anzahl von Messwerten (\(n<\infty\)). Wir ziehen also aus der theoretischen Grundgesamtheit eine Stichprobe. Für diese Stichprobe können wir eine empirische Verteilungfunktion berechnen und darstellen. Dabei stellen wir meist fest, dass diese z.B. schon ab \(n=100\) anfängt, der theoretischen Verteilungsfunktion zu ähneln. Daher gibt es Methoden, um mit einer gewissen Irrtumswahrscheinlichkeit von der empirischen Verteilung auf die theoretische zu schließen. Dies kann dann genutzt werden, um die Werte der empirischen Verteilung zu interpolieren, vor allem aber um diese in die Extrembereiche zu extrapolieren.
Anleitungen
Histogramm
Allgemeines Vorgehen
- Teile deinen Wertebereich in mehrere Klassen, indem du Klassengrenzen (breaks) definierst
- Zähle wie viele Werte jeweils zwischen den Grenzen liegen
-
Stelle die Anzahl der in dieser Klasse liegenden Messwerte auf eine der folgenden Weisen in einem Balkendiagramm dar:
- Die Balken stellen die absolute Klassenhäufigkeit, also die Anzahl der Messwerte in dieser Klasse dar
- Die Balken stellen die relative Klassenhäufigkeit, also Anzahl/Gesamtanzahl dar
- Die Balken stellen die Höufigkeitesdichte in dieser Klasse dar, also die relative Klassenhäufigkeit geteilt durch die Klassenbreite. Auf diese Weise können auch unregelmäßige Klassengrenzen gewählt werden.
R-Funktion
In R kann ein Histogramm mit dem Befehl hist erzeugt werden:
hist(temp)Die Klassen (breaks) können frei definiert werden. Mit freq=F wird nicht mehr die Anzahl der Messwerte (“Frequency”), sondern die Häufigkeitsdichte (“Density”) dargestellt.
Klassen=seq(5, 30, by=2)
hist(temp, breaks=Klassen, freq=F)Empirische Dichtefunktion
Die Dichtefunktion sozusagen ein Histogramm mit unendlich kleiner Klassenbreite. Bei Datensätzen mit begrenzter Anzahl an Messwerten bekommt ein Histogramm jedoch Lücken, wenn die Klassenbreiten zu schmal gesetzt werden. Daher verwendet die Funktion density in R eine Glättungsfunktion (z.B. einen gleitenden Mittelwert), um die Dichtefunktion darzustellen.
plot(density(temp))Empirische Verteilungfunktion
Allgemeines Vorgehen
- Enferne alle fehlenden Einträge aus dem Datensatz.
- Berechne die Anzahl der Messwerte \(n\).
- Sortiere alle Messwerte der Größe nach.
- Weise jedem Messwerte einen Plotindex \(i\) zu nach dem Muster \(i=1,2,...,n\).
-
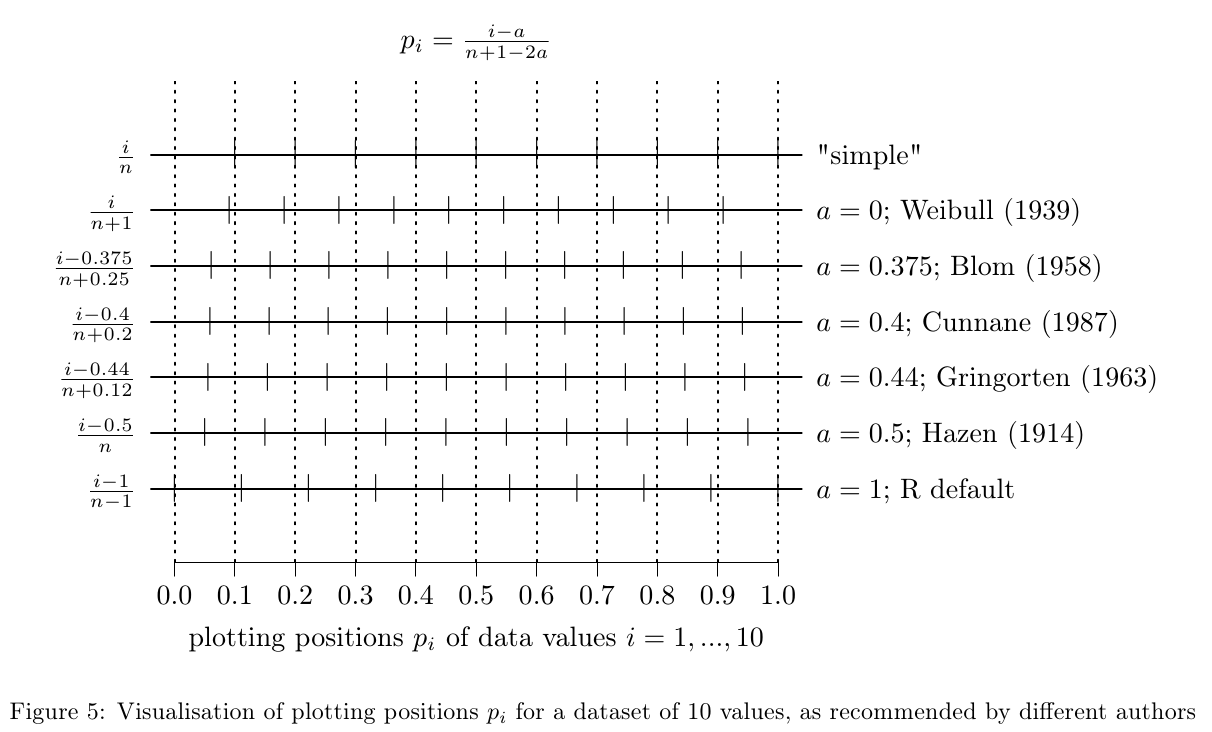
Berechne die Plotting Position \(p_i\) nach der allgemeinen Formel
\(p_i = \frac{i-\alpha}{n+1-2\alpha}\)Wähle \(\alpha\) dem Einsatzzweck entsprechend nach den Empfehlungen verschiedener Autoren (siehe nächste Abbildung). Die Unterschiede in diesen Methoden ist vor allem bei kleinen Stichproben wichtig. Bei großen Datensätzen macht die Wahl der Methode keinen großen Unterschied.
- Stelle auf der y-Achse die Plotting Positinen \(p_i\) dar und auf der x-Achse die sortierten Messwerte.
Umsetzung in R
temp <- na.omit(temp)
n <- length(temp)
i <- 1:n
p_i <- i/(n+1) # hier nach der Methode von Weibull (1939)
plot(sort(temp), p_i)Es gibt auch eine Funktion, die die empirische Veteilungsfunktion (empirical distribution function, ecdf) darstellt:
plot(ecdf(temp))Fragestellungen
Aufgaben
Erzeugen Sie für die Messgrößen “NIEDERSCHLAGSHOEHE”, “ET0” (Grasreferenzverdunstung) und “KWB” (klimatische Wasserbilanz) ein Histogramm, sowie eine empirische Dichtefunktion und Verteilungskurve für die folgenden Teildatensätze: Messwerte aus dem Monat Juli und Messwerte aus dem Monat Januar.
Stellen sie die Histogramme in getrennten Abbildungen dar. Zeichnen Sie die Dichtefunktion als Linie in das Histogrammen (Tipp: das funktioniert nur, wenn das Histogramm die Häufigkeitsdichte darstellt).
Benutzen sie lines, um die Verteilungsfunktion beider Monate in einer Abbildung darzustellen. Erstellen Sie eine zusätzliche Abbildung, die das gleiche mit der Dichtefunktion macht.
Beantworten Sie damit die Fragen:
- Regnet es weniger im Sommer?
- Haben wir in Cottbus im Winter stärkere Niederschlagsereignisse?
- In welcher Jahreszeit gibt es mehr Niederschlagsereignisse mit mittlerer Intensität?
- Warum ist es im Sommer in Cottbus trockener als im Winter?
Umsetzung
Die Abfrage von Messwerten aus einem bestimmten Monat machen Sie auf folgende Weise:
# Beispiel Abfrage des Index von Messwerten des Monats April:
index <- month(Cottbus$MESS_DATUM)==4Mit diesem Index können Sie dann die Werte abfragen, z.B. so:
tempApril <- Cottbus$LUFTTEMPERATUR[index]Wenn Sie das probieren möchten, können Sie nicht nur die Verteilungsfuktionen der Monate Juli und Januar in einer Grafik einzeichnen, sondern die Verteilungsfunktionen aller Monate. Benutzen Sie dazu eine Schleife nach dem folgenden Muster:
for(Monat in 1:12){
index <- month(Cottbus$MESS_DATUM)==Monat
# Frage Werte ab
# Berechne Verteilungsfunktion
if(Monat==1){
# Plotte Verteilungsfunktion
plot(...)
}else{
# Füge Linie der nächsten Verteilungsfunktion zu Plot hinzu
lines(...)
}
}Die nächste Übung in dieser Reihe ist die Übung zur Anpassung von theoretischen Verteilungsfunktionen (Dauerlinie)